3-4 |
低線量放射線被ばくの影響解明に迫る
|
 |
||
|
 |
||
|
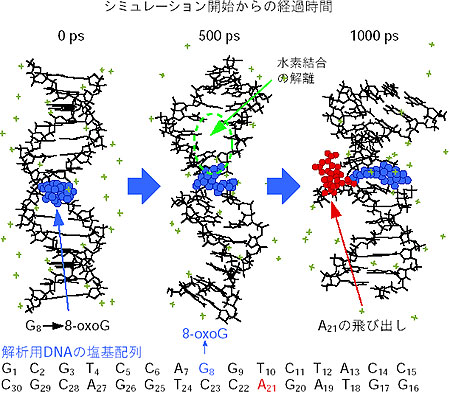
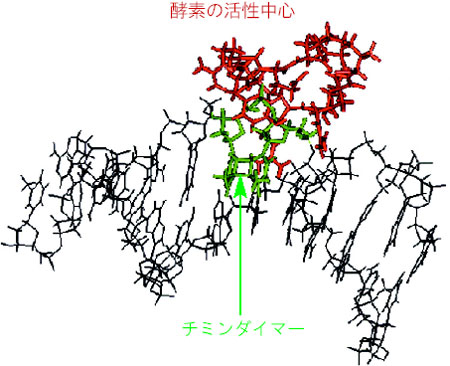
| 放射線によってDNAが傷つくと、4つの核酸塩基(チミンT、アデニンA、グアニンG、シトシンC)が変化したり、DNA鎖が切断されたりします。しかし、これらの損傷のほとんどは、細胞内にある酵素の働きで、元通りに修復されます。酵素がDNA損傷を修復する時に、どのようにして長いDNA分子の中から損傷部位を見つけ出すのでしょうか。これまでの生物実験から、酵素が認識するための信号がDNAにあること、酵素にはDNA 結合部位があってそこで損傷を認識すること、酵素とDNAの選択的な結合は酵素のアミノ酸とDNAの塩基との間の水素結合やファンデアワールス力で起こることが分かっています。しかし、酵素が選択的にDNAの損傷部位をどのようにして認識するのか、その根本的なメカニズムは良く分かっていません。 そこで、分子動力学的シミュレーションを用いて、数百ピコ秒の時間内で起こるDNAの塩基損傷と酵素との相互作用を調べました。塩基損傷としては、突然変異や発ガンの原因となる8-オキゾグアニン、皮膚がんの原因となるチミンダイマーについて検討しました。その結果、8-オキソグアニンが生じた時には、相補的塩基の間で水素結合のネットワークが壊れ、2重らせんの開裂が生じるとともに、塩基損傷が起きた反対側のDNA鎖で塩基の飛び出しが起こり、らせん構造が歪むことが分かりました(図3-11)。一方、チミンダイマーが生じると、損傷部位を中心にしてDNAらせん構造に鋭い折れ曲がりが起こることが分かりました(図3-12)。また、塩基損傷が生じない場合には、DNAの2重らせん構造に顕著な構造変化が起こらないので、酵素はDNAと結合しません。したがって、このような塩基損傷周辺で起こる2重らせん構造の変化を酵素が認識して、安定な複合体を形成するものと思われます。このDNAの構造変化に伴って起こる静電エネルギーの変化を計算した結果、損傷部分に特異的なエネルギー変化が起こることが分かりました。すなわち、修復酵素がDNA表面を走査して損傷を探す時に、この静電エネルギーの変化が損傷部位を非損傷部位とを区別する1つの重要な目印になっていることが明らかになりました。 |
| ●参考文献 M. Pinak, Computational Determination of Radiation Damage Effects on DNA Structure, Cent. Eur. J. Phys., 1(1), 179 (2003). |
| ご覧になりたいトピックは左側の目次よりお選びください。 |  |
| たゆまざる探究の軌跡−研究活動と成果2003 Copyright(c) 日本原子力研究所 |