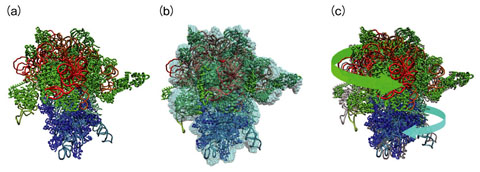
図4-24 異なる手法で明らかにされたリボソームの立体構造
近年、抗生物質に耐性のある細菌が増えているため、新しい抗生物質の開発が急務になっています。多くの抗生物質は、細菌の細胞内小器官であるリボソームで行われるタンパク質合成を阻害してその増殖を抑制します。したがって、抗生物質を効率的に開発するためには、タンパク質合成を行っている状態のリボソームの立体構造を原子レベルで明らかにすることが重要です。
タンパク質などの生体分子の立体構造は、主にX線結晶構造解析という実験手法によって調べられています。この手法は解像度が高く、タンパク質分子を構成する個々の原子の位置まで決定できますが、タンパク質の結晶化が必須で、それには様々な条件が要求されます。したがって、特定の状態にあるタンパク質の構造を明らかにすることは容易ではありません。リボソームのように多数のタンパク質や核酸から成る複合体(生体超分子)であればなおさらです。現在、いくつかの状態のリボソームの立体構造(図4-24(a),(c))が明らかにされていますが、その活動全体をとらえるには情報が少なすぎます。
近年、X線結晶構造解析とは違う、電子顕微鏡を使った生体分子の立体構造の決定が盛んに行われています。これは、電子顕微鏡により様々な方向から撮影した生体分子の二次元画像から三次元立体構造を構築する手法です。構築された立体構造の解像度が低いため、分子を構成する原子の位置を決定することは困難ですが、結晶化の必要がないため、様々な状態の分子の構造を決定することが比較的容易というメリットがあります。リボソームについてもタンパク質合成途中の様々な状態での三次元立体構造(図4-24(b))が多数決定されています。
そこで私たちは、X線結晶構造解析で得られた原子レベルの構造を変形することによって、電子顕微鏡で明らかになった構造にうまくフィットする原子モデル構造(図4-24(b),(c))を構築する計算手法を開発しました。 これにより、電子顕微鏡では構造が分かっていても、X線結晶構造解析では計測できていない状態のリボソームについても、原子モデル構造を構築できるようになりました。この手法を適用して、電子顕微鏡で調べられた様々な状態のリボソームの立体構造について原子モデル構造を構築しました。さらに、構築した原子モデル構造を統計的に解析して、タンパク質合成の際のリボソームの立体構造変化を明らかにすることに成功しました。
このような立体構造情報は、新しい抗生物質の開発に道を開くことが期待できます。