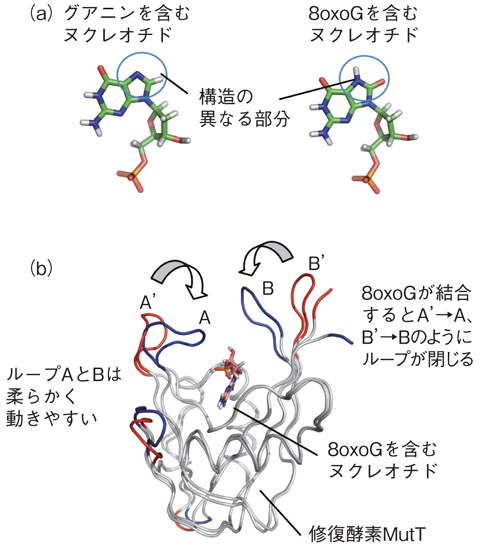
図8-18 グアニンと8oxoG及び修復酵素MutTの構造
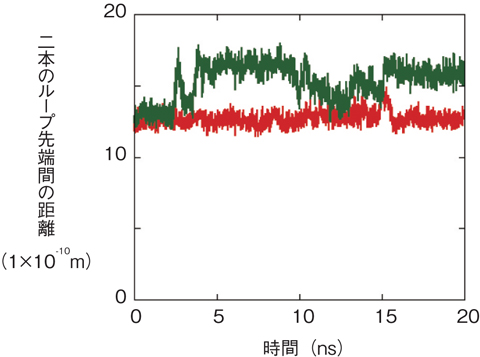
図8-19 グアニン,8oxoGと結合したときの二本のループの先端間の距離の時間変化
細胞に放射線を照射するとDNAの材料となるヌクレオチドの塩基部分が酸化損傷を起こすことがあります。この酸化したヌクレオチドがDNAに取り込まれると、細胞の突然変異等を引き起こす原因になります。そこで生物は酸化したヌクレオチドがDNAに取り込まれる前に、酵素を用いて除去しています。
塩基の酸化損傷のひとつである8-オキソグアニン(8oxoG)は図8- 18(a)に示すように正常なグアニンと分子構造の違いがほとんどありません。一般的に、酵素は結合する相手とぴったりと構造がはまることで強く結合し、構造が似ていると区別がつきにくくなります。8oxoGを持つヌクレオチドを取り除く酵素MutTも、グアニンと8oxoGの両者と結合することができますが、構造の差から予想できる以上に大きな差で強く8oxoGと結合し、効率良く除去します。この結合力の差はどのような仕組みで表れるのでしょうか。
酵素MutTはループで構成されている二本の腕を持ち、図8- 18(b)の赤の構造で示すように、何も結合していないときには開いた状態になっています。一方で、8oxoGと結合すると図8- 18(b)の青の構造で示すように8oxoGを二本の腕で取り囲むような閉じた構造になることが実験により分かっています。私たちは8oxoGとMutT及びグアニンとMutTの結合状態の分子動力学シミュレーションを行いました。
その結果、図8- 19の赤のグラフで示すように、8oxoGが結合した場合は閉じた構造のまま変化がなく、MutTの構造揺らぎも小さなままでした。しかし、図8- 19の緑のグラフが示すように、グアニンが結合した場合は二本の腕がやや開いて構造揺らぎも大きくなり、閉じた構造では不安定になります。更に詳細な構造を調べると、8oxoGとMutTの結合時に存在する特定の水素結合がグアニンとの結合時にはないことが原因で、MutTの腕の構造にずれが生じ、そのずれが次々と伝わり二本の腕の部分がきれいに閉じなくなっていました。この研究により、結合する分子の小さな構造の違いによって、その分子との結合時の安定性に大きな差が生じることが分かりました。この安定性の差が効率良い損傷部分の除去につながっていると考えられます。このように、DNAの修復機構を分子的な側面から理解することが、放射線による生物効果を解明するために必要であると考えられます。